卷积神经网络算法改进思维导图
U367456671
2024-05-24

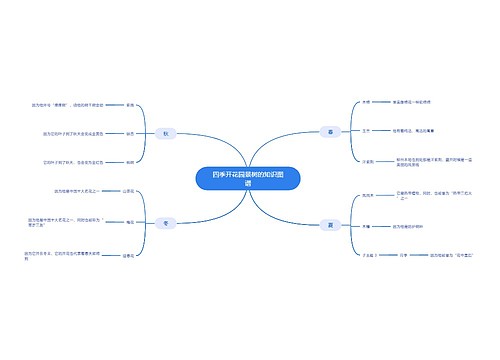
卷积神经网络算法改进简述
树图思维导图提供《卷积神经网络算法改进》在线思维导图免费制作,点击“编辑”按钮,可对《卷积神经网络算法改进》进行在线思维导图编辑,本思维导图属于思维导图模板主题,文件编号是:95c211965461f876ade3cbca181792ec
思维导图大纲
相关思维导图模版
地下水污染与防治课程思政建设总体设计流程思维导图
 U678949074
U678949074树图思维导图提供《地下水污染与防治课程思政建设总体设计流程》在线思维导图免费制作,点击“编辑”按钮,可对《地下水污染与防治课程思政建设总体设计流程》进行在线思维导图编辑,本思维导图属于思维导图模板主题,文件编号是:2a2ad0a837add4ac76b83eae2b31982c

《被算法操控的生活:重新定义精准广告、大数据和AI》思维导图
 U377992573
U377992573树图思维导图提供《《被算法操控的生活:重新定义精准广告、大数据和AI》思维导图》在线思维导图免费制作,点击“编辑”按钮,可对《《被算法操控的生活:重新定义精准广告、大数据和AI》思维导图》进行在线思维导图编辑,本思维导图属于思维导图模板主题,文件编号是:0c91e73ff7775b398b574ad797b0c912